Analyses fonctionnelles
Le service d’Analyses Fonctionnelles s’adresse aux porteurs de projets de :
- Protéomique : analyse différentielle, analyse globale, recherche de partenaires, phosphoprotéome
- Génomique : puces, RNAseq, miRNome
-
Métabolomique
Seul pré-requis : les données doivent être traitées au niveau statistique.
La formation et la mise en autonomie sur logiciels des porteurs de projet est privilégiée.
L’approche classique en matière d’analyse fonctionnelle « omic » consiste à réaliser des enrichissements dont il existe 2 grands types :
-
l’ORA : Over-Representation Analysis
Permet de savoir si dans un jeu de n protéines ou de gènes d’intérêt (préalablement sélectionné(e)s), il y a plus d’éléments impliqués dans un terme (pathway, fonction biologique, maladie, compartiment cellulaire…) que ce à quoi on serait en mesure d’attendre.
Seule une liste d’identifiants est requise.
-
Le GSEA : Gene Set Enrichment Analysis
Permet de savoir si les protéines/gènes les plus exprimé(e)s ou les moins exprimé(e)s d’un jeu de données sont impliqué(e)s de manière préférentielle dans un terme (voie canonique, fonction moléculaire…).
Pas de sélection préalable (selon la p-value, la q-value ou le ratio), les valeurs d’expression sont requises.
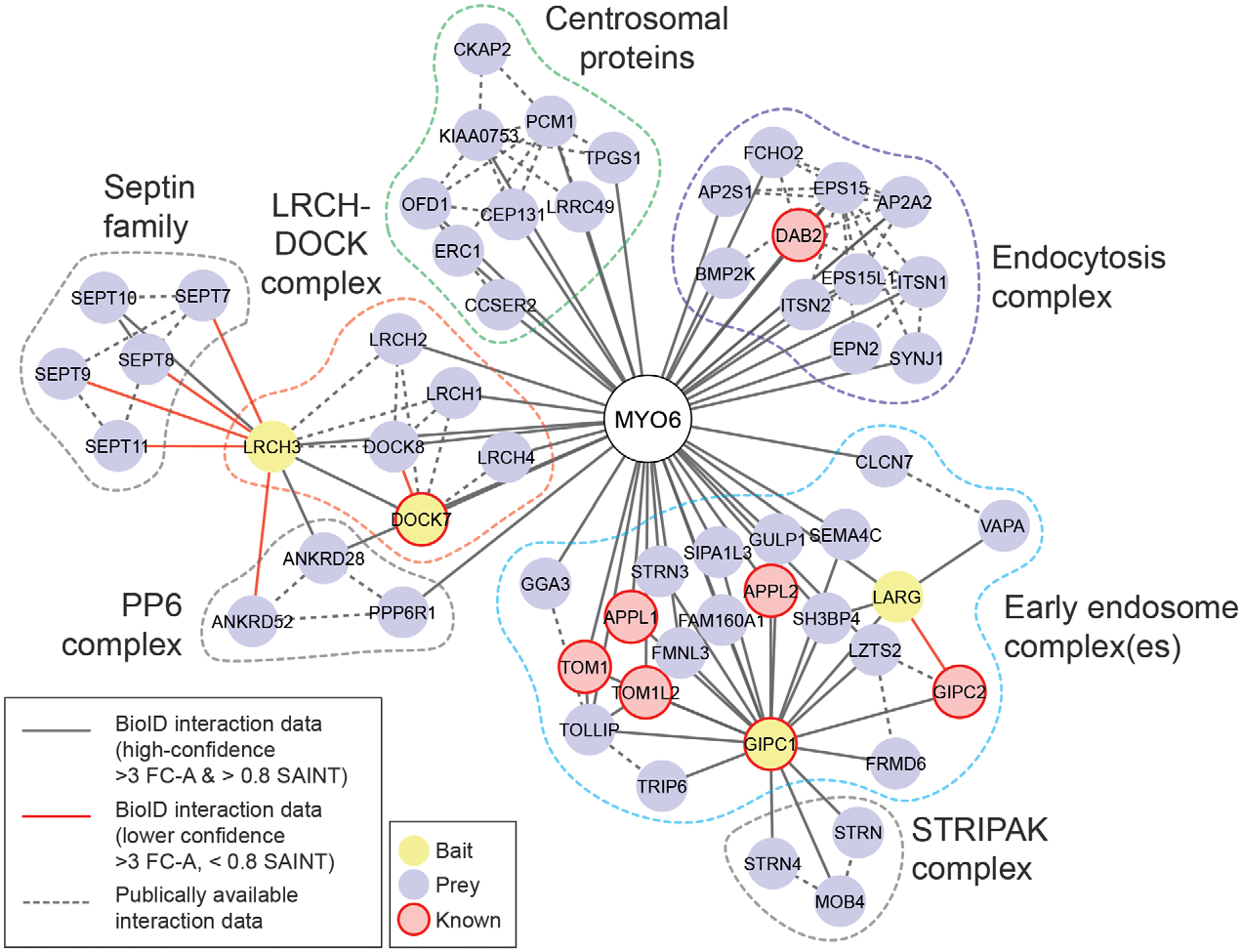
Une autre approche consiste à réaliser des réseaux d’interactions : voir si les protéines d’intérêt interagissent entre elles et déterminer quels sont les processus biologiques sous-jacents.
Les logiciels dédiés
Le service a souscrit une licence Ingenuity Pathway Analysis (IPA) de Qiagen pour ses performances et sa facilité d’utilisation.
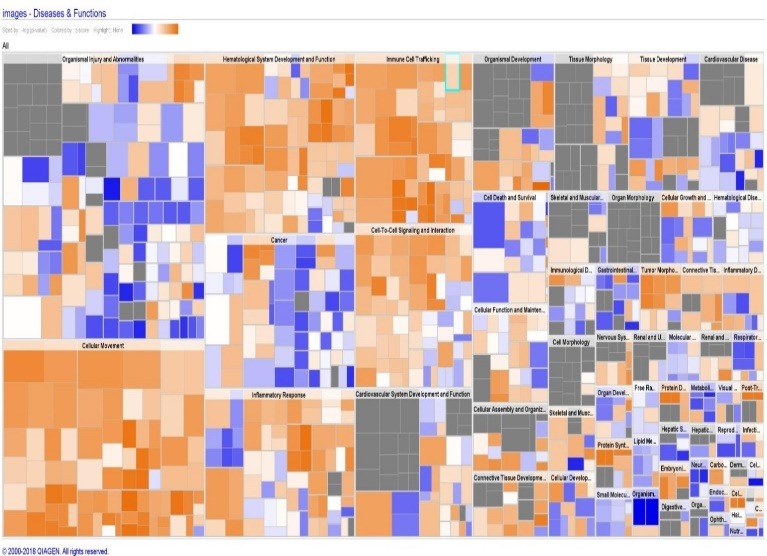
IPA exploite l’enrichissement de type « over-representation analysis » pour donner un sens biologique à une liste de transcrits ou de protéines différentiellement exprimé(e)s.
IPA identifie ainsi les voies de signalisation, les voies métaboliques, les fonctions biologiques les plus enrichies dans une analyse comparative.
IPA identifie également des régulateurs causals pouvant moduler plusieurs protéines différentielles..
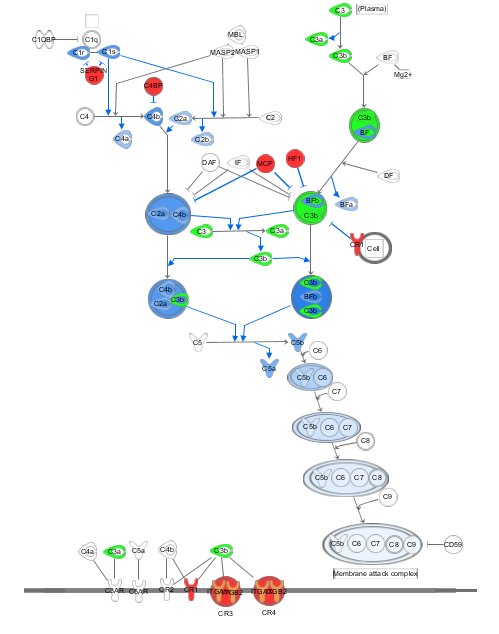
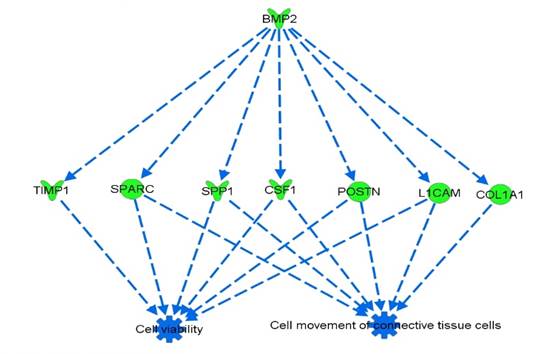
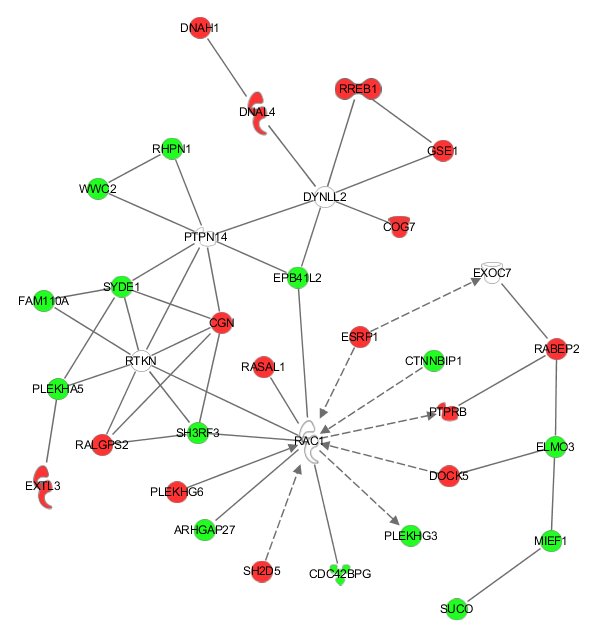
IPA génère aussi des réseaux d’interactions en connectant des protéines d’intérêt de manière directe ou indirecte par le biais de « hub ».
Le service propose également toute une gamme de solutions logicielles gratuites en ligne pour vous accompagner de l’analyse à la réalisation de réseaux, diagrammes et figures pour vos publications :
- DAVID, PANTHER, FunRich, Enrichr, GeneTrail2, ClueGO : logiciels d’ORA
- GSEA, PSEA Quant : logiciels procédant à du Gene/Protein Set Enrichment Analysis
- String : génération de réseaux d’interaction
- Cytoscape : génération de réseaux à façon
- Reactome : sa base de données d’icônes permet de réaliser de nouveaux pathways
- Perseus : réalisation d’«heat-map » et de « hierarchical clustering »…
- Crapome et REPRINT : des logiciels dédiés aux expériences d’Immuno-Précipitation : Identification de potentiels faux interactants & réalisation de réseaux d’interactions appât – proies
Thomas O’Loughlin et al 2018
Retrouvez la présentation du service d’Analyses Fonctionnelles sur ce poster.
Contact : Morgane Le Gall – 01.40.51.64.35
Contact
Morgane Le Gall
01.40.51.64.35
À lire aussi
Certification ISO9001 et Certification NFX 50-900
La plateforme Proteom’IC d'Université Paris Cité est certifiée ISO 9001 depuis octobre 2012. Proteom’IC poursuit son engagement dans une démarche d’amélioration continue en étant passée à la version 2015 de la norme depuis octobre 2018. Proteom’IC est également...

Formation « Méthodes d’analyse protéomique comparative globale »
Cette formation vous permettra de comprendre les méthodes de protéomique utilisées pour identifier et quantifier les protéines, d'apprécier l’intérêt et la puissance des techniques présentées ainsi que leurs limites et de savoir interpréter et analyser ces données de...